研究テーマ
「植物の成長・分化」を遺伝子から個体まで幅広く研究
植物は光を光合成のエネルギー源としてだけではなく、外界を知るためのシグナルとしても利用している。植物が光受容体を介して受け取った光情報をどのようにその成長・分化の制御に利用しているのか、その仕組みを遺伝子や情報分子の改変を通して解明する。

野生型のシロイヌナズナ(左)と
花成遅延変異体(右)
植物ペプチドホルモンに関する研究
植物体が成長していく過程では、体を構成する一つ一つの細胞が増殖・分化することによって全体としての形がつくられる。この時、細胞同士は多数の情報物質をやり取りして互いの振る舞いを調節し合っている。このような情報物質として古典的には少数の植物ホルモンが知られていたが、近年の生化学・遺伝子・ゲノム研究から、ペプチド性のホルモンが多数発見され、植物の体中で細胞間の様々な情報伝達を担っていることが分かってきた。
このような細胞間の情報伝達をより深く解明するため、シロイヌナズナに加えてゼニゴケというモデル植物を用いた研究を進めている。ペプチドホルモンやその受容体遺伝子を改変すると、これらの植物にはどのような影響が見られるのだろうか。さらに、情報分子や薬剤を利用することで、遺伝子改変だけでは困難な植物生理現象の操作ができないだろうか。このような観点で研究を行っており、遺伝子組み換え手法の整っていない非モデル植物への適用にも関心を持っている。
植物の概日時計、花芽形成時期の制御とユビキチン
植物の概日リズムは18世紀初頭にフランスの天文学者ド・メランによってインゲン葉の就眠(昼夜)運動として初めて報告されたとされている。最近のマイクロアレーを用いた研究では、シロイヌナズナの30-40%程度の遺伝子が約24時間の周期を持って発現変動していることが示されている。多くの植物は、自らの生物時計によるリズム(概日リズム)と外部環境の光条件を照らし合わせることでその花芽形成(花成)時期(いつ花を咲かせるか)を決めており、この概日リズムと花芽形成時期の制御にはユビキチン-プロテアソーム系が深く関わっている。
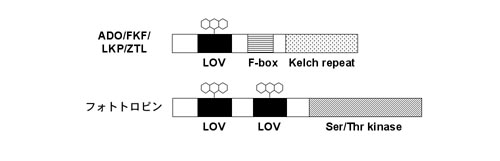
F-boxタンパク質としてのADO/FKF/LKP/ZTLファミリータンパク質
モデル高等植物であるシロイヌナズナの全ゲノム塩基配列は2000年に発表され、ユビキチン-プロテアソーム系によるタンパク質分解に機能するF-boxタンパク質遺伝子が700個以上アノテーションされている。この中には、オーキシン、ジベレリン、エチレン、ジャスモン酸といった植物ホルモンのシグナル伝達に関わるTIR1、SLY、EBF1/2、COI1や、花の形態形成に関わるUFO等が含まれている。その他に、概日時計や花芽形成(花成)時期の制御に関わるF-boxタンパク質としてADO/FKF/LKP/ZTLファミリーが知られている。これらは、N末端からLOV(Light, Oxygen, or Voltage)ドメイン、F-box、kelchリピートから構成されるタンパク質であり、シロイヌナズナには対応する3つの遺伝子(ADO1/LKP1/ZTL, ADO2/LKP2, ADO3/FKF1)が存在している(図1)。
LKP2、ZTLと概日リズム
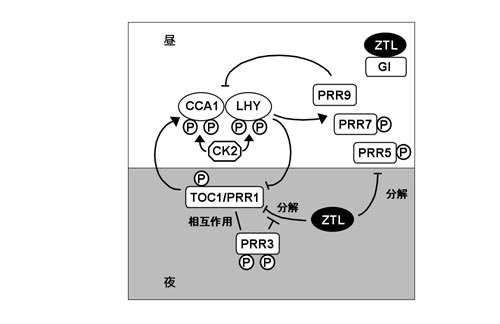
植物では、葉の就眠運動や花弁の開閉等、我々が肉眼で観察することが出来る現象だけではなく、様々な遺伝子の発現制御にも概日時計が関わっていることが知られている。シロイヌナズナの概日リズムは、おおまかに言えば、概日時計の中心振動体であるCCA1, LHYとTOC1のフィードバック制御を中心にして発生すると考えられている。MYB様転写制御因子をコードしているCCA1, LHY遺伝子は、TOC1によって発現が促進され、そのmRNA量は明け方に最大となる。他方、TOC1はプロモーター領域にCCA1, LHYが結合することで発現が抑制され、そのmRNA量が夕方から夜に最大に達する。TOC1はDNA結合領域を持たないことから、他の転写因子と相互作用してCCA1, LHY遺伝子の転写を正に制御すると考えられている。TOC1はPRRファミリーの一つで、PRR1とも呼ばれる。PRRファミリーの他の因子PRR3、PRR5、PRR7、PRR9も概日リズムの制御において中心振動体に近い役割を担っていると考えられている(図2)。
LKPファミリーの1つであるZTLの機能欠損点変異体(ztl-1, 2)では、標的タンパク質の認識に機能するkelchリピート上の保存されたアミノ酸に置換があり、概日時計の出力側であるCAB2遺伝子(早朝にmRNA量が最大となるmorning gene)の発現周期が3、4時間程度長くなり、CCR2遺伝子(夕方にmRNA量が最大となるevening gene)の発現周期や子葉の運動も長周期に変化している。機能欠損変異体とは対照的に、ZTLを過剰発現させた形質転換体ではその発現量に応じてCAB2プロモーター制御下にあるLUCレポーター遺伝子の発現周期が短くなったことから、ZTLは量依存的に概日時計を制御していると考えられている。
ZTLのLOVドメイン(LOV)は、光受容能を持つ点で非常にユニークである。LOVは、タンパク質間相互作用に関わるPAS (Per-ARNT-Sim)ドメインのサブセットの1つで、特に光,酸素,電位の変化に関与するものに共通した領域である。シロイヌナズナでは、LKPファミリーの他に、光屈性の青色光受容体であるフォトトロピン(phot)がLOVを有している(図1)。LOVにはFMN (flavin mononucleotide)等のフラビンが結合し、青色光照射によりフラビンとLOV内で保存されたシステイン残基との共有結合が誘起され,暗黒下で解離する(暗反転)。photではこのLOVの光反応により,タンパク質キナーゼ活性が制御されることが示されている。LKPファミリーのLOVはphotのLOVには無い短いループを持ち,光生化学的な性質が異なることが示されており、FKF1のLOVはアンチパラレルな方向で結合することで2量体形成に関与することが示唆されている。
近年、ZTLのLOVドメインと巨大な核タンパク質であるGIが青色光依存的に相互作用することが報告された(図2)。ZTLのmRNA量は概日時計による制御を受けず一定だが、タンパク質量は夕方にピークを示す概日リズムを示す。このZTLの蓄積リズムにはGIの関与が示唆されている。ZTLとGIは相互作用すると互いに安定化し、giではZTLの夕方の蓄積量の増加が認められない。
TOC1及びPRR5タンパク質の蓄積量にもリズムが認められるが,このTOC1及びPRR5の分解はZTLが担っている (図2)。TOC1及びPRR5とZTLは酵母2-hybrid(Y2H)、免疫共沈降により相互作用が示され、ZTLのLOVドメインがTOC1及びPRR5との結合領域である。しかし現在までZTLとTOC1との結合に青色光による影響は認められていない。野生型では、TOC1タンパク質は夕方から夜にかけて、PRR5タンパク質は昼に蓄積するが、ztl変異体では常に高いレベルで蓄積する。野生型の細胞抽出液を用いたin vitro実験でTOC1及びPRR5タンパク質は速やかに分解されるが、ztl抽出液では分解されない。そして、この分解はプロテアソーム阻害剤によって阻害される。ZTLによるTOC1及びPRR5タンパク質の分解は暗黒下において顕著に促進する。ZTLとTOC1の相互作用はPRR3が競合的にTOC1と相互作用することで阻害される (図2)。PRR3タンパク質はTOC1タンパク質とほぼ同時期に蓄積しており、TOC1の安定化に寄与していると考えられている。ZTLとGIが光依存的に相互作用することでZTLの蓄積リズムが確立され、ZTLによるTOC1及びPRR5の分解によってTOC1及びPRR5の蓄積リズムの振幅が高まることで、結果として概日時計の周期と位相が正常に保たれると考えられている。
LKP2もY2HによりTOC1及びPRR5との相互作用が示されている。また、免疫共沈降法によりGIとの相互作用が示されている。LKP2の過剰発現体では連続明、連続暗下でのCAB2、CCR2遺伝子の発現と子葉の運動の概日リズムが消失し、長日条件下で花芽形成が著しく遅延する。これらはZTL過剰発現体の形質と非常に良く似ている。これらのことからLKP2は概日時計制御に関わっていると考えられる。
概日リズムとタンパク質リン酸化酵素
中心振動体CCA1を用いたY2HによりCK2制御(α)サブユニットCKB3が単離されている。CK2は2つの触媒(β)サブユニットと制御サブユニットからなるヘテロ4量体のSer/Thrタンパク質キナーゼで、シロイヌナズナにはその構成因子としてCKA1、CKA2、及び、CKB1、CKB2、CKB3がある。いずれのCKBもCCA1と相互作用することがY2Hと組換えタンパク質を用いたin vitroの実験で示されている。また、CKA1がCCA1をin vitroでリン酸化すること、CKB1はこのCKA1によるCCA1リン酸化活性を増進することも明らかにされている。LHYも、CCA1と同様に、CK2によるリン酸化を受ける (図2)。
シロイヌナズナ形質転換体を用いた実験では、CK2を過剰発現すると中心振動体CCA1、LHY,また出力側の遺伝子であるCCR2,Lhcb,CATのmRNA蓄積量の概日周期が短くなる、赤色光によるLhcb発現誘導が弱くなる、長日、短日条件下で花芽形成が早くなり、特に短日下では顕著である、といった表現型が認められる。一方、CCA1の被リン酸化アミノ酸残基を置換した変異型CCA1を過剰発現させると、野生型CCA1を過剰発現させた植物体で見られるような概日リズムの消失は認められない。これらの結果は、CK2によるCCA1のリン酸化がCCA1の中心振動体としての機能に重要であることを示している(図2)。CCA1のリン酸化はLhcb1*3プロモーターへのDNA結合活性を促進するが、そのリン酸化がCCA1のユビキチン化とそれに続く分解の指標となっている可能性については今後の課題である。
もう一つの概日時計中心因子であるTOC1とそのファミリーであるPRR3、PRR5、及びPRR7もリン酸化される。TOC1は特に暗黒期にほとんどがリン酸化型となる。ZTLは、TOC1とPRR5それぞれのリン酸化型、非リン酸化型いずれとも相互作用するが、リン酸化型TOC1及びPRR5との相互作用のほうがやや強い。TOC1とPRR3の相互作用は、両方がリン酸化されていることを必要とする(図2)。暗黒期初期にはリン酸化されたTOC1とPRR3が相互作用し、安定化していると考えられる。PRR3はin vitro実験系においてWNK1によりリン酸化されることが報告されている。PRR5もWNK1と相互作用するが、リン酸化されるかどうかは明らかではない。TOC1をリン酸化する酵素はまだ知られていない。
FKF1と花芽形成時期
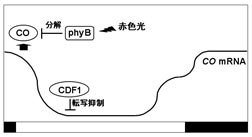
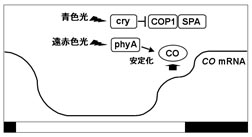
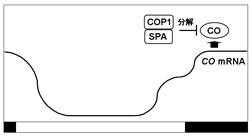
シロイヌナズナは許容的な長日植物であり、長日条件下で花成が促進される。この光周期による花成で重要な役割を果たしている遺伝子の1つにCONSTANS(CO)がある。COは長日条件下で遅咲きとなる変異体の原因遺伝子で、zinc finger型の転写制御因子をコードしており、花成促進因子であるFTの転写を活性化する。COのmRNA蓄積量は概日リズム制御を受け、タンパク質はユビキチン-プロテアソーム系による修飾・分解を受けている(図3~6)。COを過剰発現させ、そのmRNA量を一定にした植物では、COタンパク質は青色、遠赤色光下で蓄積し、赤色光下では殆ど蓄積しない。この光によるCOタンパク質の安定化には青光受容体cry1、cry2、遠赤色光受容体phyAが、不安定化には赤色光受容体phyBが関与していることが変異体を用いた解析から示されている(図3、5)。COタンパク質は光質(波長)によってその安定性が制御されているが、暗所では速やかに分解される。この暗所でのCOの分解にはCOP1とSPAファミリーの関与が示唆されている(図5、6)。COP1はRINGフィンガードメイン、コイルドコイルドメイン、WD40リピートを有するタンパク質でユビキチンリガーゼ活性を持ち、SPAファミリーはキナーゼドメインとタンパク質間相互作用に関わるWD40リピートを有している。COP1とSPAファミリーは相互作用することで暗所での形態形成を制御すると考えられているが、更にCOP1とSPA1は共にCOと相互作用することが確認されており、COP1、SPAファミリーの機能欠損変異体ではCOの蓄積が認められる。なお、COP1は明所では細胞質に存在するのに対して、暗所では核に局在することが知られている。
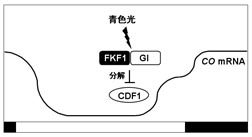
FKF1 は、LKP2、ZTLと異なり、その遺伝子発現が概日時計によって制御されている。fkf1-2では、長日条件下で野生型に見られるCO mRNAの夕方 (ZT10-16)のピークが消え、その結果、FT mRNA量が減少するため、花成が遅延する。 FKF1は青色光依存的にGIと結合する。この複合体形成はFKF1 kelchリピート領域とDof (DNA binding with One Finger)ファミリーに属する転写制御因子CDF1の結合を促進する。FKF1と結合したCDF1はユビキチン化され26Sプロテアソームにより分解されると考えられている(図4)。CDF1はCOプロモーターのDOF結合モチーフに結合してCOの転写抑制に働く(図3)。従って、CDF1の分解は、COの転写活性化とそれに続くCOタンパク質によるFTの転写活性化を引き起こし,花成が促進されると考えられている。しかし、CDF1 のRNAi植物 ではfkf1-2の花成遅延を僅かにしか回復出来ず、FKF1の標的がCDF1の他にもあると推測されている。
研究室での研究の方向性
植物の概日リズム制御やそれに関連した日長変化による花成時期制御に、ユビキチン-プロテアソーム系が深く関わっていることがここ数年で明らかにされてきている。これには、シロイヌナズナの全ゲノム塩基配列の決定とリソース整備、ルシフェラーゼレポーター系の開発といったブレイクスルーが不可欠であった。植物生理学(清末研究室)では、国内最大の植物科学研究機関である理化学研究所(RIKEN)等の外部機関と共同研究を行っており、網羅的な遺伝子発現解析や分子遺伝学的アプローチ、生化学的アプローチ、細胞生物学的アプローチ等により、LKP2の機能解析を中心に、植物の概日リズム・花成時期制御の機構解明を進めている。
プロフィール/メンバー
教授
助教
学生
- D1 高橋 剛 Takahashi Go
- B4 松田 美緒 Matsuda Mio
- B4 黒須 紀美恵 Kurosu Kimie
- B4 谷田部 綾華 Yatabe Ayaka
- B4 森内 聡美 Moriuchi Satomi
- B4 渡辺 くるみ Watanabe Kurumi
- B4 森田 修平 Morita Shuhei





