研究テーマ
微生物の天然物合成の仕組みを知り、創薬を目指す
土壌微生物である放線菌は地球上ほぼ全ての土壌に存在します。放線菌は多様な二次代謝産物を作る特徴があり、その中には抗生物質を初めとした多様な生理活性天然物も含まれます。私たちは放線菌がどのようにして生理活性物質を作り出しているのか?放線菌が生理活性物質を作ることの環境中での役割は何か?を明らかにするために研究を行っています。また、新たな医薬品開発を目指し、放線菌の作る新規生理活性物質探索に基づく創薬研究も行っています。
眠っている抗生物質生産能力を覚醒する技術の開発
一株の放線菌が生産できる二次代謝産物の数はゲノム解読から30種類以上と見積もられています。しかし、そのうち生産が確認できているものは数種類しかなく、ほとんどは遺伝子発現が極微量か全く発現していないものです。新たな創薬シーズを得るためには、放線菌のこれら眠っている遺伝子を効率的に発現させる必要があり、私たちは共培養と薬剤による活性化の二つの手法に着目して研究を展開しています。
(1)複合培養:ミコール酸含有細菌と放線菌の共培養による抗生物質生産
これまでの私たちの研究から、ミコール酸と呼ばれる脂肪酸を細胞表層に持つ細菌(ミコール酸含有細菌)と放線菌を混ぜ合わせて培養すると、放線菌がミコール酸含有細菌に刺激され、新たな抗生物質を作る現象「複合培養」が発見されました(図1)。本現象はミコール酸含有細菌と放線菌の接触刺激を介して起こります(図2)。目も耳も持たない放線菌がミコール酸含有細菌を認識し、抗生物質生産が誘導される作用機構の解明を私たちは目指しています。

図1 : 純粋培養と複合培養における赤色色素生産誘導の違い。
ミコール酸含有細菌であるTsukamurella pulmonisは放線菌Streptomyces lividansの赤色色素生産を誘導する。複合培養条件(図右)のみ赤色色素生産が見られる。これはS. lividansがT. pulmonisによる刺激を受けて、赤色色素生産を開始したためである。
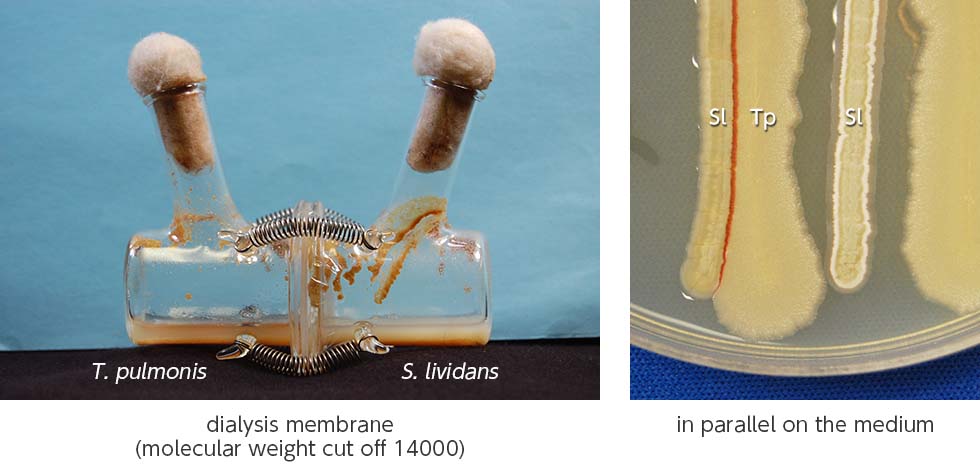
図2 物理的接触によって赤色色素生産誘導が起こる
(左)別府フラスコを用いて透析膜で両菌を分けて培養すると赤色色素生産は起こらないことから、物質のやりとりによる現象ではなく、生菌同士の物理的接触を介した現象であることが明らかとなった。(右)プレート上で両菌株が接触するように植えると、接触している部分だけ赤色色素が生産されることからも物理的接触が必要であることがわかる。
また、複合培養法を用いて、眠っている遺伝子を効率的に覚醒させる手法の確立も目指しています。これまでに、国内の大学との共同研究などにより、複合培養を用いて約40種類の新規化合物を発見しています(図3)。
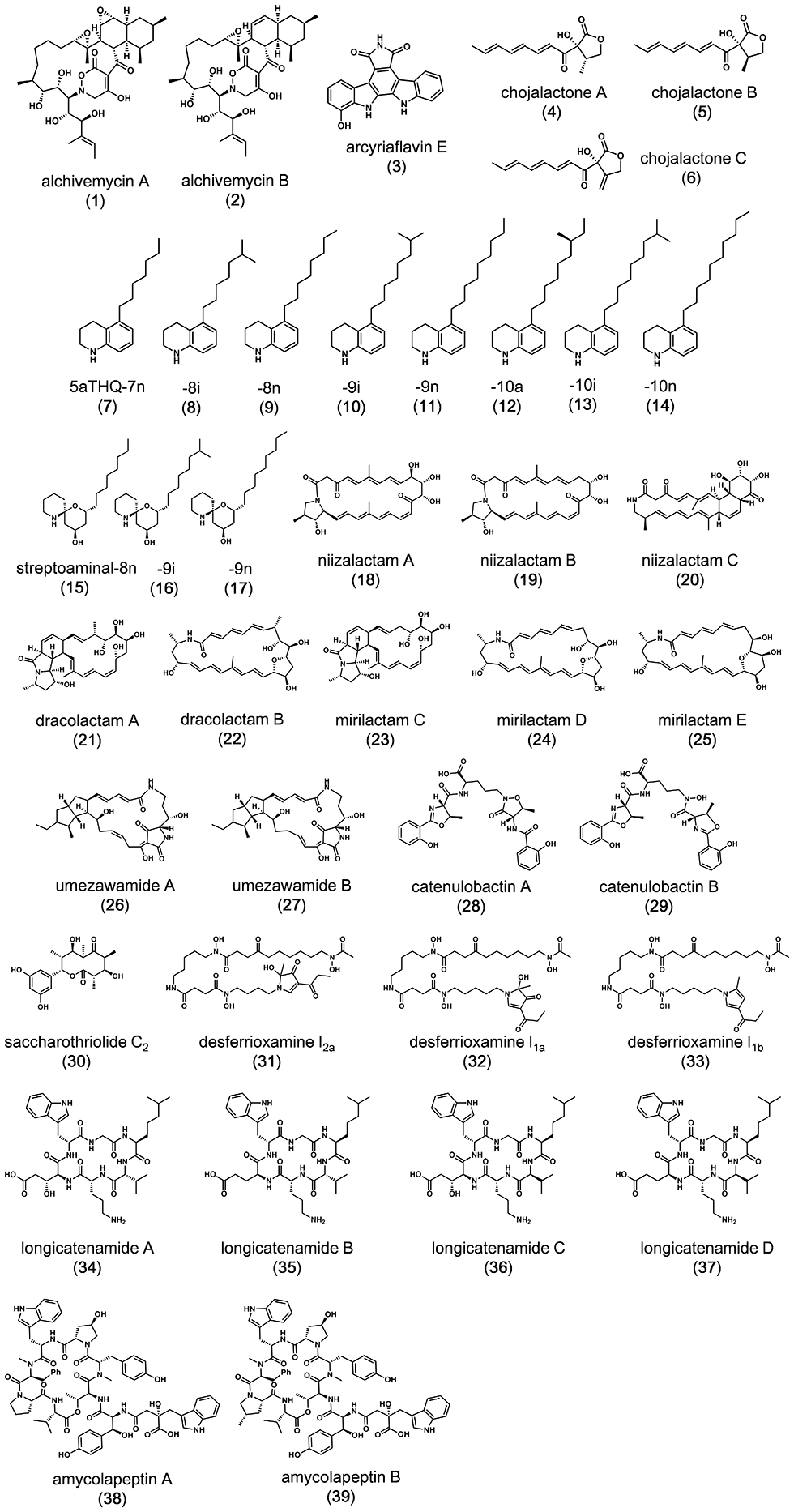
図3 これまでに複合培養法によって発見された新規二次代謝産物
(2) ゴードスポリン:放線菌の二次代謝生産を誘導する薬剤
ゴードスポリン(図4)は放線菌Streptomyces sp. TP-A0584が生産する二次代謝産物ですが、他の放線菌に与えると二次代謝生産の誘導活性を示します(図5)。ゴードスポリンによる活性化機構を分子レベルで明らかにすることにより、眠っている遺伝子の効率的な覚醒方法の確立を目指します。
![]()
図4: ゴードスポリンの化学構造、19残基からなるペプチド化合物であり、内部にアゾール環(赤、青)が6箇所とデヒドロアラニン(緑)が2箇所存在する。
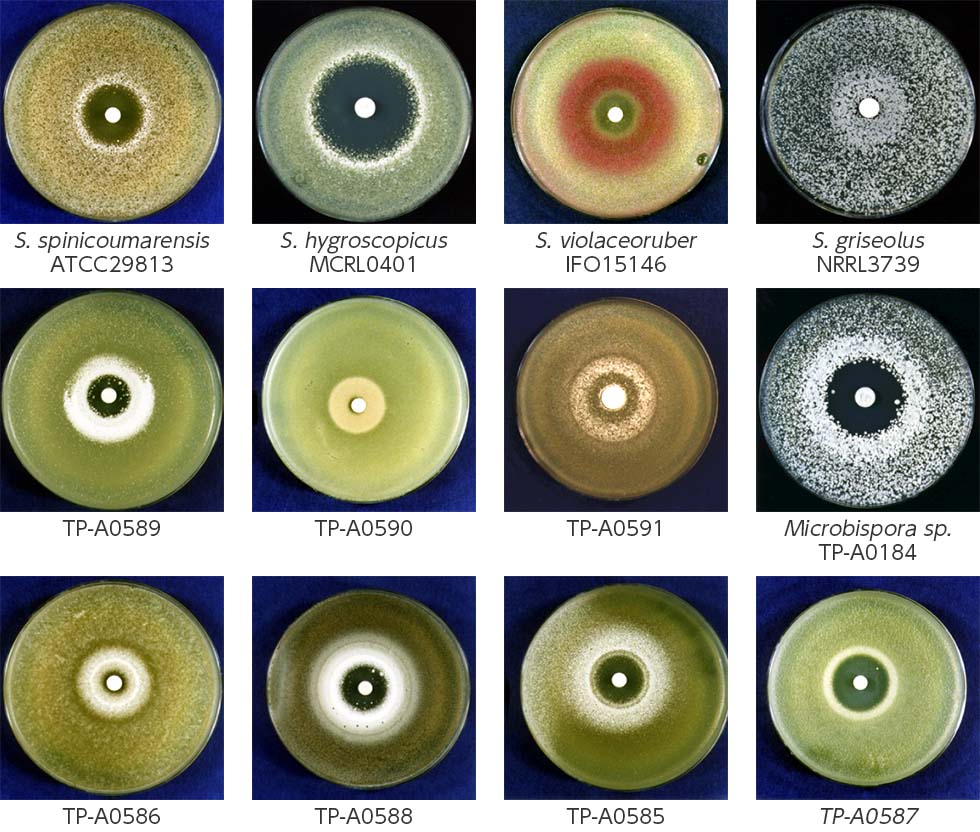
図5: ゴードスポリンが様々な放線菌に対して与える影響。ゴードスポリンを染みこませたペーパーディスクを中心にプレート一面に植えた放線菌が影響を受けているのが観察される。ゴードスポリンを使って放線菌の抗生物質生産をコントロールできる可能性がある。
中分子創薬へ向けた翻訳後修飾ペプチドの生合成研究
分子量500~2,000 の中分子化合物を用いた創薬は、抗体などのバイオ医薬品と同等の基質特異性を示し、低分子並みに安価に製造できるという利点がある。中でもペプチド系中分子天然物は発酵生産による供給が可能である事から、ペプチドの固相合成法よりも製造コストが著しく低いため、注目されている。
私たちはRiPPs(Ribosomally synthesized and post-translationally modified peptides)と呼ばれるリボソームによって翻訳合成された後に修飾酵素によってアミノ酸が修飾されるペプチド系化合物群に注目している。RiPPsは翻訳後修飾を受けているため、通常のペプチドに比べ、3次元構造を取りやすく創薬ターゲットとの高親和性を発揮する。また、ペプチダーゼなどによる分解を受けにくくなるため、細胞内で安定である利点がある。これまでに私たちはゴードスポリン、ラクタゾール、ゴードビオニン、ソラビオマイシンの4つのRiPPs化合物を発見し(図6)、生合成機構を解明し、医薬品リードとして有望な類縁体創製を目指した研究を行っている。
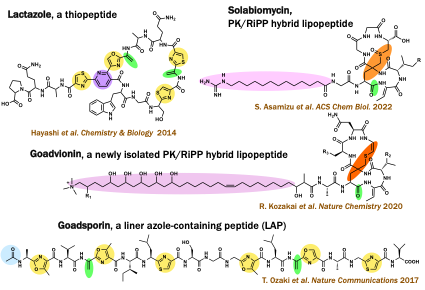
図6 私たちが研究しているRiPPs化合物群
ハイライト部分は翻訳後修飾構造を示している。これらの翻訳後修飾構造によって、化合物の生理活性や生体内における安定性が高まると考えられている。
活動実績
論文発表
2023年
1. S. Ijichi, S. Hoshino, S. Asamizu, H. Onaka. SolS-catalyzed sulfoxidation of labionin to solabionin drives antibacterial activity of solabiomycins. Bioorganic & Medicinal Chemistry Letters 89: 129323 (2023) doi: 10.1016/j.bmcl.2023.129323
2. T. Nagakubo, S. Asamizu, T. Yamamoto, M. Kato, T. Nishiyama, M. Toyofuku, N. Nomura, H. Onaka. Intracellular Phage Tail-Like Nanostructures Affect Susceptibility of Streptomyces lividans to Osmotic Stress. mSphere e0011423 (2023). doi: 10.1128/msphere.00114-23
3. Y. Lei, S. Asamizu, T. Ishizuka, H. Onaka. Regulation of Multidrug Efflux Pumps by TetR Family Transcriptional Repressor Negatively Affects Secondary Metabolism in Streptomyces coelicolor A3(2). Applied and Environmental Microbiology 89(3): e0182222 (2023) doi: 10.1128/aem.01822-22.
2022年
1. A. A. Vinogradov, Y. Zhang, K. Hamada, J.S. Chang, C. Okada, H. Nishimura, N. Terasaka, Y. Goto, K. Ogata, T. Sengoku, H. Onaka, H. Suga. De Novo Discovery of Thiopeptide Pseudo-natural Products Acting as Potent and Selective TNIK Kinase Inhibitors. Journal of the American Chemical Society, 144(44):20332-20341 (2022) doi: 10.1021/jacs.2c07937
2. S. Asamizu, S. Ijichi, S. Hoshino, H. Jo, H. Takahashi, Y. Itoh, S. Matsumoto, H. Onaka. Stable isotope-guided metabolomics reveals polar-functionalized fatty-acylated RiPPs from Streptomyces. ACS Chemical Biology 17(10): 2936-2944 (2022) doi: 10.1021/acschembio.2c00601
3. S. Asamizu, A.A.C. Pramana, S. Kawai, Y Arakawa, H. Onaka. Comparative metabolomics reveals a bifunctional antibacterial conjugate from combined-culture of Streptomyces hygroscopicus HOK021 and Tsukamurella pulmonis TP-B0596. ACS Chemical Biology 17(9): 2664-2672 (2022) doi: 10.1021/acschembio.2c00585
4. M. Yanagisawa, S. Asamizu, K. Satoh, Y. Oono, H. Onaka. Effects of carbon ion beam-induced mutagenesis for the screening of RED production-deficient mutants of Streptomyces coelicolor JCM4020. PLoS One 17(7): e0270379 (2022) https://doi.org/10.1371/journal.pone.0270379
5. A.A. Vinogradov, Chang, H. Onaka, Y. Goto, H. Suga. Accurate Models of Substrate Preferences of Post-Translational Modification Enzymes from a Combination of mRNA Display and Deep Learning. ACS Central Science 8(6): 814-824 (2022) https://doi.org/10.1021/acscentsci.2c00223
6. M. Kato, S. Asamizu, H. Onaka. Intimate relationships among soil bacteria: actinomycetes and mycolic acid-containing bacteria. Scientific Reports 12: 7222 (2022) https://doi.org/10.1038/s41598-022-11406-2
7. Y. Asai, T. Hiratsuka, M. Ueda, Y. Kawamura, S. Asamizu, H. Onaka, M. Arioka, S. Nishimura, M. Yoshida. Differential Biosynthesis and Roles of Two Ferrichrome-Type Siderophores, ASP2397/AS2488053 and Ferricrocin, in Acremonium persicinum. ACS Chemical Biology, 17(1), 207-216 (2022) https://doi.org/10.1021/acschembio.1c00867
8. N. Koyama, A. Hikima, S. Asamizu, H. Onaka, H. Zhang, H. Tomoda. Kimidinomycin, a New Antibiotic against Mycobacterium avium Complex, Produced by Streptomyces sp. KKTA-0263. The Journal of Antibiotics, 75: 72-76 (2022) https://doi.org/10.1038/s41429-021-00494-3
9. E. J. Han, S. R. Lee, S. Hoshino, M. R. Seyedsayamdost. Targeted Discovery of Cryptic Metabolites with Antiproliferative Activity. ACS Chemical Biology, 17: 3121-3130 (2022) https://doi.org/10.1021/acschembio.2c00588
10. S. M. Horwitz, T. C. Blue, J. A. Ambarian, S. Hoshino, M. R. Seyedsayamdost, K. M. Davis. Structural insights into inhibition of the drug target dihydroorotate dehydrogenase by bacterial hydroxyalkylquinolines. RSC Chemical Biology 3: 420-425 (2022) https://doi.org/10.1039/d1cb00255d
11. 星野翔太郎 コロナ禍における米国留学 未曽有のパンデミックが変えた研究生活. 化学と生物, 60(4): 205-209 (2022).
2021年
1. T. Nagakubo, T. Yamamoto, S. Asamizu, M. Toyofuku, N. Nomura, H. Onaka. Phage tail-like nanostructures affect microbial interactions between Streptomyces and fungi. Scientific Reports, 11(1): 20116 (2021) https://doi.org/10.1038/s41598-021-99490-8
2. N. Oku, S. Takemura, H. Onaka, Y. Igarashi. NMR characterization of streptogramin B and L-156,587, a non-synergistic pair of the streptogramin family antibiotic complexes produced inductively by a combined culture of Streptomyces albogriseolus and Tsukamurella pulmonis Magnetic Resonance in Chemistry 60(2): 261-270 (2021) https://doi.org/10.1002/mrc.5219
3. Y. Jiang, T. Matsumoto, T. Kuranaga, S. Lu, W. Wang, H. Onaka, H. Kakeya. Longicatenamides A-D, two diastereomeric pairs of cyclic hexapeptides produced by combined-culture of Streptomyces sp. KUSC_F05 and Tsukamurella pulmonis TP-B0596. The Journal of Antibiotics, 74(5): 307-316 (2021) 10.1038/s41429-020-00400-3
4. C. Pan, T. Kuranaga, X. Cao, T. Suzuki, N. Dohmae, N. Shinzato, H. Onaka, H. Kakeya. Amycolapeptins A and B, cyclic nonadepsipeptides produced by combined-culture of Amycolatopsis sp. and Tsukamurella pulmonis. J Org Chem. 15;86(2):1843-1849 (2021) https://doi.org/10.1021/acs.joc.0c02660
5. 尾仲宏康 ユニークな生合成機構を持つ新規リポペプチド系天然物の発見.B&Iバイオサイエンスとインダストリー,79(2): 116-117 (2021)
2020年
1. R. Kozakai, T. Ono, S. Hoshino, H. Takahashi, Y. Katsuyama, Y. Sugai, T. Ozaki, K. Teramoto, K.Teramoto, K. Tanaka, I. Abe, S. Asamizu, and H. Onaka. Acyltransferase that catalyses the condensation of polyketide and peptide moieties of goadvionin hybrid lipopeptides. Nature Chemistry, 12: 869-877 (2020) https://doi.org/10.1038/s41557-020-0508-2
2. A.A. Vinogradov, E. Nagai, JS. Chang, K. Narumi, H. Onaka, Y. Goto, H. Suga. Accurate Broadcasting of Substrate Fitness for Lactazole Biosynthetic Pathway from Reactivity-Profiling mRNA Display. Journal of the American Chemical Society, 142, 48, 20329-20334 (2020) https://doi.org/10.1021/jacs.0c10374
3. A. A. Vinogradov, M. Shimomura, Y. Goto, T. Ozaki, S. Asamizu, Y. Sugai, H. Suga, H. Onaka. Minimal lactazole scaffold for in vitro thiopeptide bioengineering. Nature Communications, 11: 2272 (2020) https://doi.org/10.1038/s41467-020-16145-4
4. A. A. Vinogradov, M. Shimomura, N. Kano, Y. Goto, H. Onaka, and H. Suga. Promiscuous enzymes cooperate at the substrate level en route to lacatzole A. Journal of the American Chemical Society, 142, 32, 13886-13897 (2020) https://doi.org/10.1021/jacs.0c05541
5. L. Vaario, S. Asamizu, T. Sarjala, N. Matsushita, H. Onaka, Y. Xia, H. Kurokochi, S. Morinaga, J. Huang, S. Zhang, C. Lian. Bioactive properties of streptomyces may affect the dominance of Tricholoma matsutake in shiro. Symbiosis, 1-13 (2020) https://doi.org/10.1007/s13199-020-00678-9
6. I. Morita, T. Mori, T. Mitsuhashi, S. Hoshino, Y. Taniguchi, T. Kikuchi, K. Nagae, N. Nasu, M. Fujita, T. Ohwada, I. Abe. Exploiting a C-N Bond Forming Cytochrome P450 Monooxygenase for C-S Bond Formation. Angewandte Chemie International Edition, 59: 3988-3993 (2020). https://doi.org/10.1002/anie.201916269
7. 尾仲宏康, 浅水俊平 物理的接触が介在する放線菌二次代謝誘導. Journal of Environmental Biotechnology(環境バイオテクノロジー学会誌)20: 13-22 (2020)
2019年以前の主要論文
1. R. Sugiyama, T. Nakatani, S. Nishimura, K. Takenaka, T. Ozaki, S. Asamizu, H. Onaka, H. Kakeya. Chemical Interactions of Cryptic Actinomycete Metabolite 5‐Alkyl‐1, 2, 3, 4‐tetrahydroquinolines through Aggregate Formation. Angewandte Chemie International Edition, 131 (38): 13620-13625 (2019) https://doi.org/10.1002/anie.201905970
2. T. Ozaki, K. Yamashita, Y. Goto, M. Shimomura, S. Hayashi, S. Asamizu, Y. Sugai, H. Ikeda, H. Suga, and H. Onaka. Dissection of goadsporin biosynthesis by in vitro reconstitution leading to designer analogs expressed in vivo. Nature communications, 8: 14207 (2017) https://doi.org/10.1038/ncomms14207
3. R. Sugiyama, S. Nishimura, T. Ozaki, S. Asamizu, H. Onaka, and H. Kakeya. Discovery and total synthesis of streptoaminals: Antimicrobial [5,5]-spirohemiaminals from the combined-culture of Streptomyces nigrescens and Tsukamurella pulmonis. Angewandte Chemie International Edition, 55(35): 10278-10282 (2016) https://doi.org/10.1002/anie.201604126
4. S. Hayashi, T. Ozaki, S. Asamizu, H. Ikeda, S. Ōmura, N. Oku, Y. Igarashi, H. Tomoda, and H. Onaka. Genome mining reveals a minimum gene set for the biosynthesis of 32-membered macrocyclic thiopeptides lactazoles. Chemistry & Biology, 21(5): 679-88 (2014) 10.1016/j.chembiol.2014.03.008
5. Arnison PG, et al., Ribosomally synthesized and post-translationally modified peptide natural products: overview and recommendations for a universal nomenclature. Natural Product Reports, 30(1): 108-160 (2013) https://doi.org/10.1039/C2NP20085F
6. H. Onaka, Y. Mori, Y. Igarashi, and T. Furumai. Mycolic acid-containing bacteria induce natural-product biosynthesis in Streptomyces species. Applied and Environmental Microbiology, 77(2): 400-406 (2011) https://doi.org/10.1128/AEM.01337-10
7. Y. Wang, H. Chen, M. Makino, Y. Shiro, S. Nagano, S. Asamizu, H. Onaka, and S. Shaik. Theoretical and experimental studies of the conversion of chromopyrrolic acid to an antitumor derivative by cytochrome P450 StaP: The biological role of water molecules. Journal of the American Chemical Society, 131: 6748-6762 (2009) https://doi.org/10.1021/ja9003365
8. Y. Wang, H. Hirao, H. Chen, H. Onaka, S. Nagano, and S. Shaik. Electron transfer activation of chromopyrrolic acid by cytochrome P450 en-route to the formation of an antitumor indolocarbazole derivative - theory supports experiment. Journal of the American Chemical Society, 130(23): 7170-7171 (2008) https://doi.org/10.1021/ja711426y
9. M. Makino, H. Sugimoto, Y. Shiro, S. Asamizu, H. Onaka, and S. Nagano Crystal structures and catalytic mechanism of cytochrome P450 StaP that produces the indolocarbazole skeleton. Proceedings of the National Academy of Sciences of USA, 104: 11591-11596 (2007) https://doi.org/10.1073/pnas.0702946104
10. H. Onaka, M. Nakaho, K. Hayashi, Y. Igarashi, and T. Furumai. Cloning and characterization of goadsporin biosynthetic gene cluster from Streptomyces sp. TP-A0584. Microbiology, 151: 3923-3933 (2005) https://doi.org/10.1099/mic.0.28420-0
11. H. Onaka, S. Taniguchi, Y. Igarashi, and T. Furumai. Characterization of the biosynthetic gene cluster of rebeccamycin from Lechevalieria aerocolonigenes ATCC 39243. Bioscience, Biotechnology and Biochemistry, 67: 127-138 (2003) BBB論文賞受賞論文 https://doi.org/10.1271/bbb.67.127
12. H. Onaka, S. Taniguchi, Y. Igarashi, and T. Furumai. Cloning of the staurosporine biosynthetic gene cluster from Streptomyces sp. TP-A0274 and its heterologous expression in Streptomyces lividans. The Journal of Antibiotics, 55: 1063-1071 (2002) https://doi.org/10.7164/antibiotics.55.1063
13. H. Onaka, H. Tabata, Y. Igarashi, Y. Sato and T. Furumai. Goadsporin, a chemical substance which promotes secondary metabolism and morphogenesis in streptomycetes. I. Purification and Characterization. The Journal of Antibiotics, 54: 1036-1044 (2001) https://doi.org/10.7164/antibiotics.54.1036
14. H. Onaka, N. Ando, T. Nihira, Y. Yamada, T.
Beppu and S. Horinouchi. Cloning and characterization of the A-factor receptor
gene from Streptomyces griseus. Journal of Bacteriology
177: 6083- 6092 (1995) doi: 10.1128/jb.177.21.6083-6092.1995
プロフィール/メンバー
教授
尾仲 宏康 (Hiroyasu Onaka)

教授経歴
1993年 東京大学農学部農芸化学科卒業
1998年 東京大学大学院農学生命科学研究科博士課程修了
農学博士(東京大学,博農第1903号)
1997年 日本学術振興会特別研究員
1999年 富山県立大学工学部生物工学研究センター 助手
2006年 富山県立大学工学部生物工学研究センター 講師
2010年 富山県立大学工学部生物工学科 准教授
2012年 東京大学農学部微生物潜在機能探索(発酵研究所)寄付講座 特任教授
2017年 東京大学農学部微生物潜在酵素(天野エンザイム)寄付講座 特任教授
2023年 学習院大学理学部生命科学科 教授
助教

星野 翔太郎(Shotaro
Hoshino)
助教経歴
2014年 東京大学薬学部薬科学科卒業
2016年 日本学術振興会 特別研究員 DC1
2019年 東京大学大学院薬学系研究科博士後期課程修了
2019年 日本学術振興会 海外特別研究員
2019年 米国プリンストン大学化学科 博士研究員
2022年 東京大学大学院農学生命科学研究科 特任助教
2023年 学習院大学理学部生命科学科 助教
学生
D1 伊地知 新太
B4 星野 千優
B4 道下 ひかる
B4 久保木 綾梨
B4 福原 彩穂
B4 三浦 颯斗

2023年4月


