【研究成果・プレスリリース】謎に包まれていたカイコガ科昆虫2種の W染色体配列の完全解読に成功 〜性染色体進化の解明の第一歩〜
2024.06.21
謎に包まれていたカイコガ科昆虫2種のW染色体配列の完全解読に成功〜性染色体進化の解明の第一歩〜
ポイント
- モデル生物であるカイコおよびイチジクカサンの雌ゲノム配列を過去最高の精度で決定しました。
- カイコのW染色体は90%が反復配列で構成されており、従来アセンブリ(ゲノム配列を計算機で復元すること)が不可能であると考えられてきましたが、最新のロングリードシーケンシング技術(PacBio HiFi reads, Bionano Optical genome mapping, Nanopore ultralong reads)を用いて、2種のW染色体配列を1本にアセンブリすることに成功しました。
- カイコおよびイチジクカサンのW染色体は、Z染色体が異型化して生じた染色体であることが判明しました。
- カイコの雌決定遺伝子であるFemがトランスポゾン(ゲノム中の可動性因子)の融合によって偶然に誕生した遺伝子であることが明らかになりました。
- さまざまなカイコ系統、特に間性形質を示す系統のW染色体配列の解読および比較解析が行われることが期待されます。

研究の概要
学習院大学理学部生命科学科の李允求助教、同嶋田透教授、岩手大学の藤本章晃特別助教(現所属:九州大学大学院)、同佐原健教授、基礎生物学研究所の山口勝司主任技術員、同重信秀治教授、国立遺伝学研究所の豊田敦教授らの研究グループは、最新のロングリードシーケンシング技術を用い、これまで配列決定が不可能であるとされてきたカイコのW染色体を含む、雌ゲノムアセンブリの構築および、カイコと同じくカイコガ科に属するイチジクカサンの雌ゲノムアセンブリの構築に成功しました。この成果は、カイコガ科における性染色体の進化を理解する一助となります。
本研究成果は、2024年6月13日(日本時間)に国際学術誌Molecular Ecologyのオンライン版に掲載されました。
本研究はJSPS科学研究費助成事業(J18H03949)、2022 PacBio APAC Plant & Animal Sciences HiFi-SeqXP Grant co-hosted by NovogeneAIT、基礎生物学研究所共同利用研究、学習院大学計算機センター共同研究の支援を受けて実施されました。
研究の背景
カイコなどの鱗翅目昆虫は、性染色体として雄がZ染色体を2本、雌がZ染色体とW染色体を1本ずつ持つ雌ヘテロ型のグループです。鱗翅目昆虫のW染色体は謎に包まれた染色体であり、その起源は何か、どれくらいの遺伝子が存在しているか、ほとんど明らかになっていません。特に、モデル生物であるカイコのW染色体からは、これまでにFem※1と呼ばれる雌性決定遺伝子が単離されたのみであり、そのほかの遺伝子の存否はおろか、その配列・構造すら未決定なのが現状です。
W染色体に関する研究が進みにくい原因の一つが、配列解読の難しさにあります。鱗翅目昆虫の雌では減数分裂時に遺伝的組換えが起こらないため、W染色体には変異が蓄積しやすく、さらにトランスポゾンなどの反復配列が集積しやすいという特徴があるからです。
2019年になって、米国Pacific Bioscience(PacBio)社からHigh Fidelity(HiFi)readsという高精度の配列解読技術が発表されました。研究グループは、HiFi readsを用いることによって、鱗翅目昆虫のW染色体、特に、そのほとんどが反復配列に占領されていると予想されている、カイコのW染色体配列を決定できるのではないかと考えました。
研究の内容
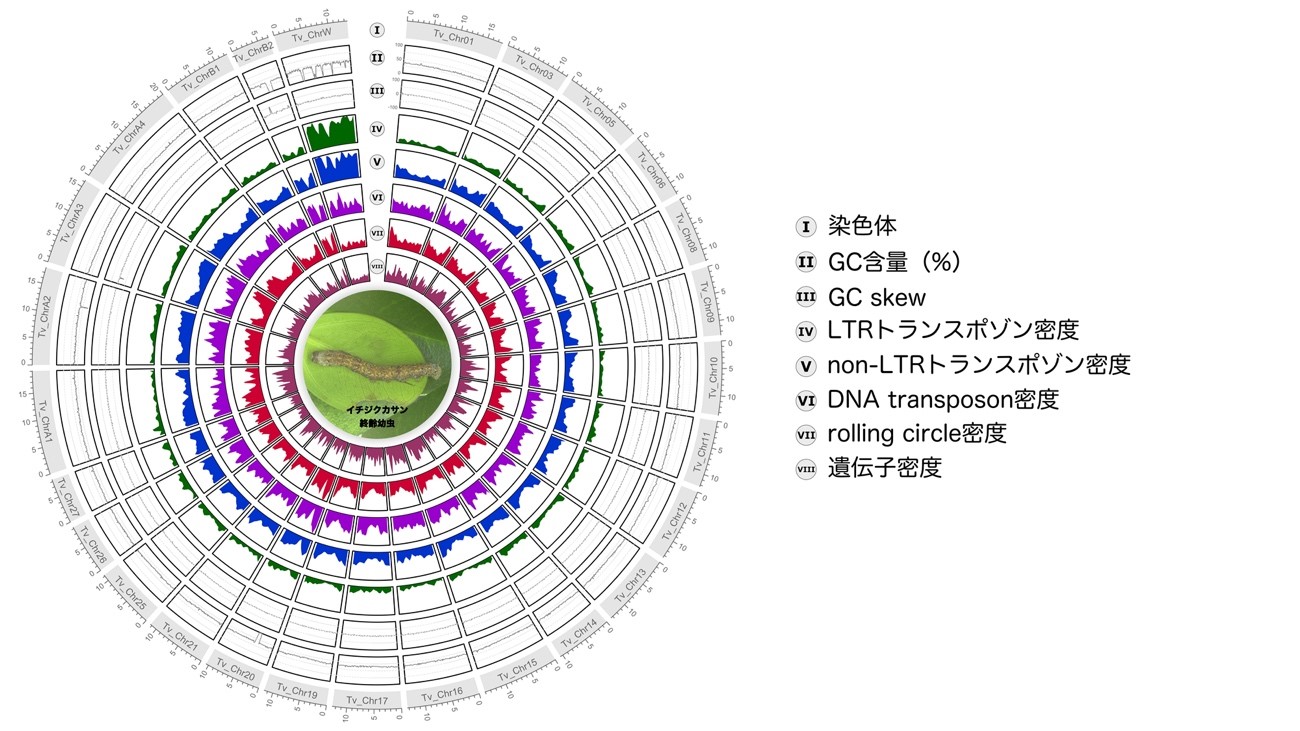
カイコと、比較対照としてカイコと同じくカイコガ科に属するイチジクカサンの雌ゲノム解読を実施しました。カイコに関してはPacBio HiFi readを用いてデータを取得しました。イチジクカサンに関してはOxford Nanopore Ultra-Long read※2を用いてデータを取得しました。続けて、Bionano Saphyrを用いたオプティカルゲノムマッピング※3によるスキャフォールディング※4を行いました。その結果、W染色体を含めた全ての染色体について、それぞれ1本の配列にまでスキャフォールディングすることに成功し、米国National Center for Biotechmology Information(NCBI)のデータベース※5に登録しました。研究グループが登録したカイコ、イチジクカサンのゲノムアセンブリはリファレンス配列※6として認証され、現在最も高精度なゲノムアセンブリの一つという評価を受けています。
注目すべきは、W染色体の構造です。カイコは、従前の予測通り、その90%近くが反復配列に占領された染色体であることが判明しました。カイコのW染色体上の反復配列の種別割合は、常染色体およびZ染色体のそれとは大きく様相が異なっており、LTR型レトロトランスポゾン※7がW染色体において大きな割合を占めていることも明らかになりました。また、イチジクカサンのW染色体も、カイコのW染色体ほど極端ではないにせよ、Z染色体や常染色体とは反復配列の種別割合が異なっており、イチジクカサンのW染色体でもLTR型レトロトランスポゾンの割合が多いことがわかりました。
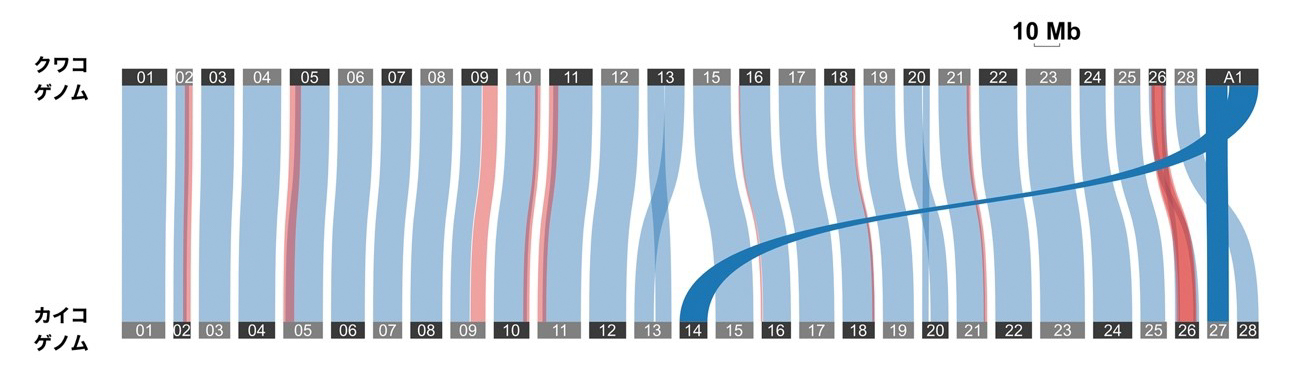
興味深いことに、カイコのW染色体とイチジクカサンのW染色体は、ぞれぞれのZ染色体と、塩基配列レベルで全く相同性を示しません。さらに2種のW染色体同士の比較においても、同じカイコガ科のW染色体であるにもかかわらず、塩基配列レベルでは全く相同性がありませんでした。しかし、W染色体から発現している遺伝子を丹念に調べてみると、Z染色体上に座乗する遺伝子と相同性があることがわかりました。このことは、両種のW染色体がかつてはZ染色体だったことを示唆しています。
雌性決定遺伝子Femについても、驚くべき事実が判明しました。カイコのW染色体上に、全長が保存されているFemの配列は129コピーも存在し、痕跡的なものも含めるともっと数は多いことがわかりました。さらに、イチジクカサンのW染色体にはFemは存在せず、Femに依存的な雌性決定機構は、カイコガ科の中でもカイコに近縁な限られた種(クワコなど)だけが採用しているむしろ珍しい性決定機構であることが明らかになりました。また、Femはその配列的な相同性から、Z染色体上に存在する雄性決定遺伝子であるMascの対立遺伝子※8なのではないかと考えられてきましたが、イチジクカサンとカイコのW染色体の配列比較により、Femは、トランスポゾンが複数融合して偶然に生じた配列である蓋然性が高いということが判明しました(図)。この推論は、先述の「Fem依存的な性決定機構を持つのは、カイコとそのごく近縁種に限られる」という、2014年のFemの発見以降、十年にわたる世界の鱗翅目研究者たちの報告を裏付けるものです。
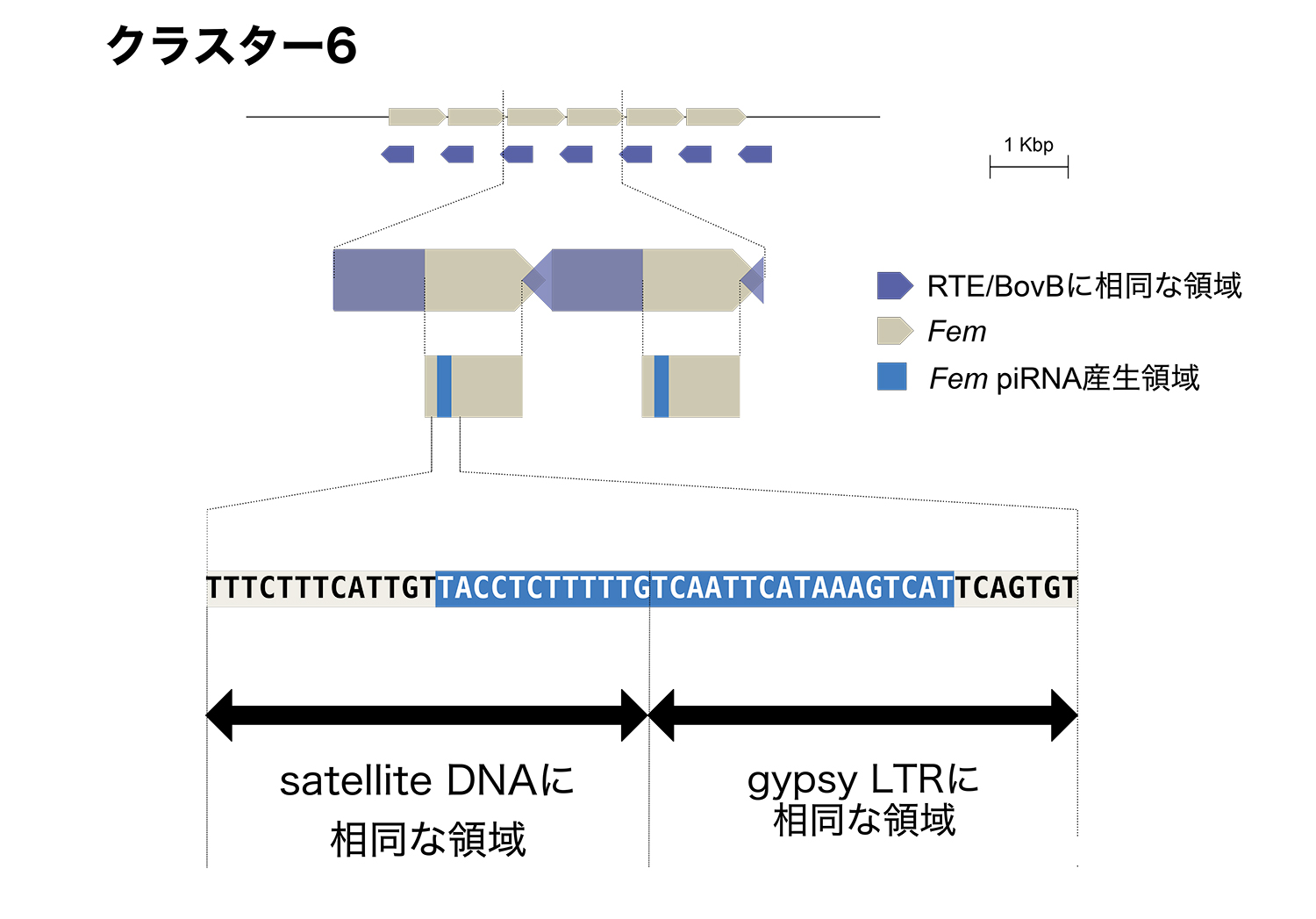
図 Femを構成するトランスポゾンを示す。例としてクラスター6のFemを図示した。Femコピーを跨ぐようにしてRTE/BovB(LINEの1種)と相同な領域が存在する。Fem piRNA産生領域(雌性決定に重要な領域)は、第8染色体上のsatellite DNAおよびgypsyのLTRに相同性を示すことが判明した。
今後の展開
W染色体の配列が明らかになったことから、遺伝子ノックインなどの技術を用いることで、雌性特異的なゲノム編集が可能になったといえます。また、研究グループが構築したW染色体配列を基にして、さまざまなカイコ系統、特に間性形質を示す系統のW染色体配列の解読および比較解析が行われることが期待されます。イチジクカサンは、カイコガ科の進化において、最も早く共通祖先から分岐したグループであり、 W染色体の比較解析を行うには、やや遠縁だったかもしれません。そのため、イチジクカサンとカイコの中間に位置するような種(テンオビシロカサンやウスバクワコなど)においてW染色体配列が決定されることで、より良い解像度でW染色体の進化をトレースできるようになると期待されます。
用語解説
※1 Fem
2014年に発見されたカイコの雌性決定遺伝子。Femはタンパク質コード遺伝子ではなく、Fem mRNAはpiRNA経路に認識されてpiRNA前駆体として振る舞う。Fem mRNAから生じるpiRNAをFem piRNAと呼び、Fem piRNAは雄性決定遺伝子Mascと相補な配列を有する。W染色体を持つ個体では、Fem piRNAとMasc mRNAの配列相補性をアンカーとして、Fem piRNAと複合体を形成しているPiwiタンパク質がMasc mRNAにリクルートされ、Masc mRNAを切断し不活性化する。Mascが発現しないと雄化が起きないため、その個体は雌になる。ちなみに、piRNAとはPIWI-interacting RNAのことを指し、24-31塩基程度の小分子RNAである。
※2 Oxford Nanopore Ultra-Long read
Oxford Nanopore Technologies社が提供するロングリードシーケンシング技術によって得られたロングリードのこと。HiFi read以前のPacBio社のロングリードであるContinuous long read(CLR)よりも長い配列を一度に決定することができるが、エラー率はCLRよりも高いとされる。
※3 オプティカルゲノムマッピング
高分子ゲノムDNAを蛍光標識し、Chipと呼ばれるナノチャネルアレイの中で電気泳動を行い、DNA1分子ごとに泳動像を撮影する手法。蛍光のシグナルパターンをもとに、ドラフトアセンブリのコンティグ同士をスキャフォールディング※4することができる。
※4 スキャフォールディング
ショートリードデータ、あるいはロングリードデータから一次的に作成された(ゲノム)アセンブリのことをドラフトアセンブリと呼ぶ。ドラフトアセンブリが染色体スケールであることは稀であり、通常はドラフトアセンブリに含まれる配列(コンティグと呼ぶ)をさらにつなぎ合わせる作業を要する。この作業のことをスキャフォールディングと呼ぶ。スキャフォールディングの結果、コンティグ同士が繋がって生じた、より長い配列のことをスキャフォールドと呼ぶ。
※5 National Center for Biotechmology Information(NCBI)のデータベース
ここでは、NCBI datasetsのことを指す。カイコおよびイチジクカサンのゲノムアセンブリには、それぞれGCA_030269925.2、GCA_030269945.1というアクセッション番号が割り振られている。
※6 リファレンス配列
ある生物種のゲノム配列として、基準とされている配列、またはそのゲノムアセンブリのことを指す。NCBIに登録されているゲノムアセンブリは冗長であり、例えばカイコに関していえば9種類のゲノムアセンブリが登録されている。その中で、もっとも精度が高いアセンブリがリファレンス配列として認証を受ける。
※7 LTR型レトロトランスポゾン
トランスポゾンは、DNA型トランスポゾンとレトロトランスポゾンに大別され、レトロトランスポゾンは、その配列的特徴から、LTR型レトロトランスポゾンと非LTR型レトロトランスポゾンに大別される。LTRはLong terminal repeatの略であり、LTR型レトロトランスポゾンは自身の配列の末端に数百塩基から数千塩基長のリピート配列を有する。
※8 対立遺伝子(アレル)
同じ遺伝子座に複数の種類がある時、それらを対立遺伝子と呼ぶ。カイコのW染色体はZ染色体に起源を持つので、これらの染色体は異型化が極端に進んでいるものの、相同染色体と見做すことができる。MascとFemは互いに一部分だけ相同な配列を有するため、W染色体の配列解読以前からFemはMascのアレルだと考えられることもあった。
論文情報
論文名:W chromosome sequences of two bombycid moths provide an insight into the origin of Fem
雑誌名:Molecular Ecology
著者名:Jung Lee, Toshiaki Fujimoto, Katsushi Yamaguchi, Shuji Shigenobu, Ken Sahara,Atsushi Toyoda, Toru Shimada
URL:https://doi.org/10.1111/mec.17434
DOI:10.1111/mec.17434
発表者
李允求 学習院大学理学部生命科学科 助教
藤本章晃 岩手大学農学部植物生命科学科 特別助教:研究当時
現:九州大学大学院農学研究院 学術研究員
山口勝司 基礎生物学研究所超階層生物学センター 主任技術員
重信秀治 基礎生物学研究所超階層生物学センター 教授
佐原健 岩手大学農学部植物生命科学科 教授
豊田敦 国立遺伝学研究所ゲノム・進化研究系 特任教授
嶋田透 学習院大学理学部生命科学科 教授