【研究成果】キメラリボソームの作製と特性理解によるリボソームタンパク質の進化の考察
2021.09.30
キメラリボソームの作製と特性理解によるリボソームタンパク質の進化の考察
Evolution of Ribosomal Protein S14 Demonstrated by the Reconstruction of Chimeric Ribosomes in Bacillus subtilis
発表者:
| 赤沼元気 | 学習院大学理学部生命科学科・助教 |
| 河村富士夫 | 千葉大学・客員教授 |
| 渡辺智 | 東京農業大学・准教授 |
| 渡辺正樹 | 東京農業大学・修士 |
| 大川典哉 | 東京農業大学・修士 |
| 名取陽祐 | 立教大学・博士 |
| 七宮英晃 | 福島県立医科大学・准教授 |
| 朝井計 | 東京農業大学・教授 |
| 千葉櫻拓 | 東京農業大学・教授 |
| 吉川博文 | 東京農業大学・教授 |
| 相馬亜希子 | 千葉大学・講師 |
| 菱田卓 | 学習院大学・教授 |
| 山田康之 | 立教大学・教授 |
発表のポイント:
○枯草菌のリボソームに他のバクテリアのリボソームタンパク質を組み込んだキメラリボソームを作製した。
○S14リボソームタンパク質を置換した70Sキメラリボソームの翻訳活性は野生型リボソームよりも低かった
○置換したS14の近傍に結合する他のリボソームタンパク質の30Sリボソームへの結合能も低下した
○リボソームタンパク質の進化には、リボソーム上で近くに結合するタンパク質の共進化も重要であることを提唱した
発表の概要:
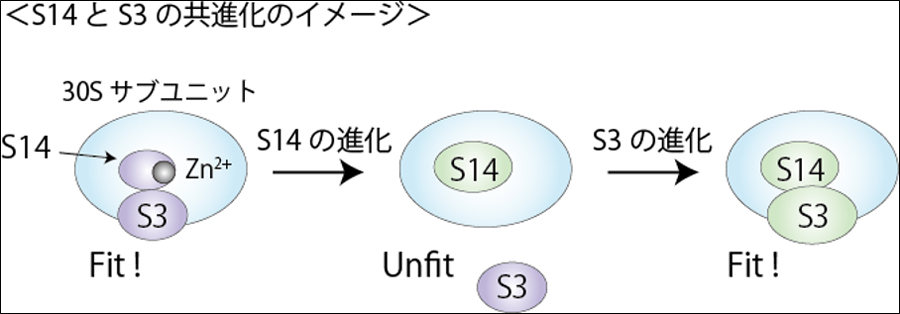
リボソームタンパク質の進化を理解する目的で、枯草菌のリボソームを形成するリボソームタンパク質の一つであるS14(S14Bs)を、大腸菌やシアノバクテリアのリボソームタンパク質(S14Ec, S14Se)に入れ替えたキメラリボソームを作製した。細胞増殖に必須なリボソームタンパク質のin vivo(生体内)での完全置換は本研究が最初の例である。キメラリボソームを持つ枯草菌の細胞増殖速度は低下しており、S14EcとS14Seが枯草菌リボソームに完全に適合できていないことが考えられた。詳細な解析から、70Sキメラリボソームの翻訳活性は野生型リボソームと比較して低いこと、リボソーム上でS14の近傍に結合するリボソームタンパク質であるS3とS2の30Sキメラリボソームに対する結合能が低いことが分かった。S14は亜鉛イオン(Zn2+)を結合するサイズの小さいタンパク質(枯草菌型)から、Zn2+に依存しないサイズの大きなタンパク質(大腸菌・シアノ型)へと進化してきたと考えられている。今回の研究から、S14の進化に伴い、S3やS2もそれに適した進化を遂げる必要があったと予想し、S14に加えてS3もシアノ型(S3Se)に入れ替えたキメラリボソームを作製した。S14SeとS3Seを含むキメラリボソームを導入した枯草菌の増殖速度は、S14SeとS3Bsを含むキメラリボソームを導入した枯草菌よりも若干早かったこと、S3Seが枯草菌リボソームで機能するためにはS14Seが必須だったことも、S14とS3の共進化の可能性を示すものである。
本成果は、「Journal of Bacteriology」に掲載されました。
(論文URLはこちら https://journals.asm.org/doi/10.1128/JB.00599-20)
本件は、学習院大学学校長裁量枠「研究力強化事業(国際学術誌論文掲載補助)」より掲載費を助成しています。
発表内容:
【研究の背景と経緯】
自然界には3種のリボソームタンパク質S14が存在する。1つ目は亜鉛イオン(Zn2+)を結合するタイプ(C+ type)で、2つ目と3つ目は亜鉛を結合せず、C+ typeよりも30アミノ酸残基程大きなC- short typeと40アミノ酸残基程大きなC- long typeである。原始的なS14はC+ typeであり機能維持にZn2+を必要とするが、Zn2+に対する依存から脱却するため、進化の過程でサイズの大きくなったC- long typeが生まれてきたと予想されている。バクテリアのリボソームは進化の過程でサイズの大きくなったS14を受け入れる必要があったと考えられるが、バクテリアリボソームはどの程度S14の変化に対して寛容なのか、S14の進化に伴いリボソームにどのような変化が必要だったのかを調査した研究は無かった。発表者らのこれまでの研究から、枯草菌はC+ typeとC- short typeの2種のS14を有し、細胞内のZn2+濃度変化に伴い2種のS14を使い分けることが分かっていた。そこで進化の途中に位置しているとも考えられる枯草菌リボソームに、大腸菌やシアノバクテリアが持つ最も進化したC- long typeのS14を人為的に導入した場合の影響を観察することでリボソームタンパク質の進化の過程を考察することを目的とした。
【研究の内容】
リボソームタンパク質S14を入れ替えるためには、枯草菌のS14をコードする遺伝子を破壊し、大腸菌やシアノバクテリアのS14遺伝子を枯草菌ゲノムに導入する必要がある。しかし、S14は枯草菌が増殖するために必須なタンパク質であるため、S14をコードする遺伝子を破壊すると枯草菌は増殖できない。そこで、人為的に発現誘導が可能な大腸菌やシアノバクテリアのS14遺伝子を先に枯草菌ゲノムに組み込み、S14EcやS14Seを発現させた条件で枯草菌S14遺伝子を破壊した。この方法で作製した枯草菌株の70Sリボソームのタンパク質構成を調べると、枯草菌S14が失われた代わりに、大腸菌やシアノバクテリアのS14が枯草菌リボソームに結合したキメラリボソームが形成されていることが確認できた。これらの枯草菌の増殖速度を比較すると、S14EcやS14Seを導入した株では増殖速度が著しく低下していた。また、S14EcやS14Seを含む枯草菌では、高い翻訳活性を持つポリソーム量が低下し、翻訳不活性な30Sと50Sサブユニット量が増加していた。これらの結果は、作製した枯草菌の細胞内翻訳活性が低下していることを示している。この理由を探るため、活性型の70Sキメラリボソームを枯草菌細胞から調製し、in vitro(試験管内)で翻訳活性を測定した結果、キメラリボソームの翻訳活性は野生型リボソームの60%以下まで低下していた。従って、翻訳活性を持つ完全な70Sであっても、S14EcとS14Seが含まれるリボソームは十分な翻訳活性を持たないことが分かった。また、キメラリボソームを含む枯草菌では翻訳不活性型の30Sと50Sサブユニットが蓄積していたことから、30Sと50Sが会合して活性型の70Sを形成する能力が低下していると考えられた。この異常に蓄積した30Sのタンパク質構成を解析したところ、S14EcとS14Seは十分に結合しているにも関わらず、リボソームタンパク質S3, S2の結合能が低下していることが判明した。S3, S2は30Sリボソーム中でS14の近傍に結合するため、サイズの大きなS14が結合することで30Sの構造が変化し、S3, S2が結合しにくくなったと考えられる。これらの結果から、進化したS14にフィットするようにS3やS2も進化を遂げたのではないかと予想し、S14に加えてS3もシアノ型に入れ替えたキメラリボソームの作製を試みた。当初、シアノバクテリアのS3(S3Se)のみを含むキメラリボソームの構築も試みたが、このリボソームを含む枯草菌は増殖できないことが分かった。そこであらかじめS14Seを導入した枯草菌にS3Seを導入したところ、増殖可能であることが分かった。さらに、S14SeとS3Seの両方を導入した枯草菌の増殖速度は、S14Seのみを導入した枯草菌よりも若干早かった。この結果は、S3Seが枯草菌で機能するためには、進化したS14(S14Se)が必須であること、S14Seには枯草菌のS3よりもシアノバクテリアのS3がフィットすることを示唆するものである。発表者らはこれらの結果から、S14とS3の共進化の可能性を提唱した。
【今後の展開】
今回の結果から、キメラリボソームの構築とその特性理解によって、リボソームタンパク質S14の進化を考察することができた。今後は様々なリボソームタンパク質を入れ替えたキメラリボソームを作製し、解析することでリボソーム全体の進化を俯瞰できるような研究を展開していきたい。
【用語解説】
リボソームについて ・・・ リボソームは遺伝子からの情報(mRNA)を読み取ってタンパク質を合成(翻訳)する複合体で、全ての生物が生きるために必須な装置である。バクテリアのリボソームは、30Sと50Sの二つのサブユニットが会合して翻訳活性を持つ70Sリボソームを構成する。翻訳活性の高いリボソームが1本のmRNAに複数結合した状態をポリソームと呼ぶ。30Sは1種のrRNAと20種程度のリボソームタンパク質から構成され、50Sは2種のrRNAと35種程度のリボソームタンパク質から構成される。今回の解析対象であるS14リボソームタンパク質は30Sを構成する14番目のタンパク質である。
研究についてのお問い合わせ先:学習院大学理学部生命科学科助教 赤沼元気
このページについてのお問い合わせ先:学習院大学学長室広報センター